Sa proseso ng pagsasagawa ng mga genetic na pag-aaral, madalas tayong nakakaranas ng hindi sapat na mga sample ng RNA, halimbawa, para sa pag-aaral ng maliliit na anatomical oral tumor, kahit na mga single-cell na sample, at mga sample ng mga partikular na mutation ng gene na na-transcribe sa napakababang antas sa mga selula ng tao.Siyempre, para sa pagsusuri sa COVID-19, kung ang mga pamunas ay wala sa tamang lugar o hindi sapat na oras sa panahon ng sampling, ang laki ng sample ay magiging napakababa, kaya naman lumabas ang Commission of Health and Family Planning dalawang araw na ang nakalipas at pumasa sa pagsusulit, at kung ang nucleic acid sampler ay hindi kumuha ng anim na sample, maaari mo itong iulat.
Ang sensitivity ng reagent ay mahalaga dahil mayroon tayong problema o problemang iyon, kaya ano ang maaari nating gawin upang mapabuti ang sensitivity ng RT-PCR?
Bago natin talakayin ang mga posibleng solusyon, banggitin natin ang dalawang malaking komplikasyon sa sitwasyong nabanggit natin.
Una sa lahat, nag-aalala kami tungkol sa pagkawala ng RNA kapag mayroon lamang kaming ilang mga populasyon ng cell sa aming sample.Kung gagamitin ang tradisyonal na paraan ng paghihiwalay at paglilinis, tulad ng paraan ng column o paraan ng pag-ulan ng nucleic acid, malaki ang posibilidad na mawala ang ilang sample.Ang isang solusyon ay ang magdagdag ng molekula ng carrier, gaya ng tRNA, ngunit kahit ganoon, walang garantiya na OK ang aming eksperimento sa pagbawi.
Kaya ano ang isang mas mahusay na paraan?Ang isang magandang opsyon para sa mga nakakulturang cell o microanatomical sample ay ang paggamit ng direktang lysis.
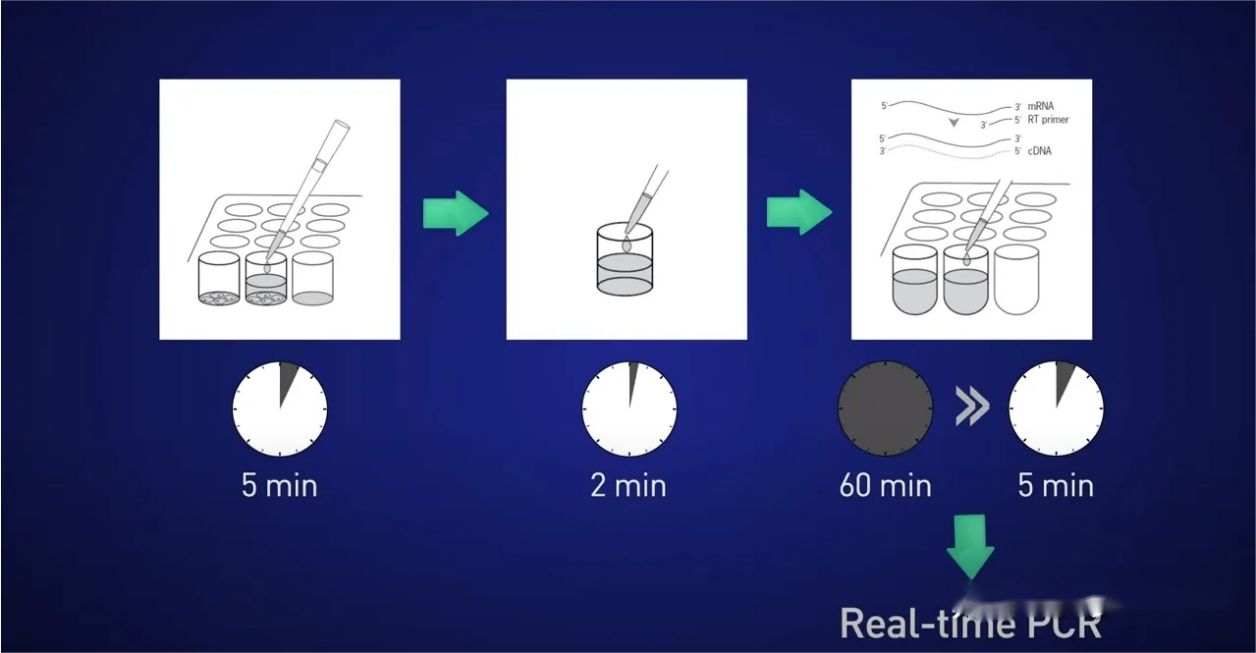
Ang ideya ay hatiin ang mga cell sa loob ng 5 minuto, bitawan ang RNA sa solusyon, pagkatapos ay itigil ang reaksyon sa loob ng 2 minuto, pagkatapos ay idagdag ang lysate nang direkta sa reverse transcription reaction upang walang RNA na mawawala, at sa wakas ay ilagay ang resultang cDNA nang direkta sa real-time na reaksyon.
Ngunit paano kung, dahil sa isang limitadong panimulang punto o isang maliit na halaga ng target na gene expression, maaari naming i-recycle ang lahat ng RNA at hindi pa rin magbigay ng sapat na mga template upang makakuha ng magandang real-time na signal?
Sa kasong ito, maaaring maging lubhang kapaki-pakinabang ang hakbang na pre-amplification.
Ang sumusunod ay isang pamamaraan upang mapataas ang sensitivity pagkatapos ng reverse transcription.Bago magsimula, kailangan nating tanungin sa ibaba ng agos kung aling mga target ang interesado tayo, upang magdisenyo ng mga partikular na panimulang aklat para sa mga target na ito para sa pre-amplification.
Ito ay maaaring makamit sa pamamagitan ng paggawa ng halo-halong primer na may hanggang 100 pares ng mga panimulang aklat at isang ikot ng reaksyon na 10 hanggang 14 na beses.Samakatuwid, kailangan ang isang Master Mix na partikular na idinisenyo para sa pangangailangang ito upang paunang palakasin ang nakuhang cDNA.
Ang dahilan ng pagtatakda ng bilang ng mga cycle sa pagitan ng 10 at 14 ay ang limitadong bilang ng mga cycle na ito ay nagsisiguro ng randomness sa pagitan ng iba't ibang mga target, na mahalaga para sa mga mananaliksik na nangangailangan ng dami ng molecular na impormasyon.
Pagkatapos ng pre-amplification, makakakuha tayo ng malaking halaga ng cDNA, upang ang sensitivity ng pagtuklas sa back-end ay lubos na mapabuti, at maaari pa nating palabnawin ang sample at magsagawa ng maraming real-time na reaksyon ng PCR upang maalis ang mga posibleng random na error.
Oras ng post: Abr-11-2023